빅데이터 분석기사 실기 관련 블로그 링크
빅데이터 분석기사 실기
팀프로젝트가 끝나고 11월 30일에 예정된 빅데이터 분석기사 실기시험을 일주일만에 벼락치기로 해보려 했건만 기출문제를 보니 맨땅에 헤딩은 아닌것같고, 시험공부와 관련돼서 검색해보니 공부에 관한 요령(?)이 잘 정리된 블로그를 발견해 링크를 남겨놓고 공부하려고 한다. 작성된 순서대로 공부할 예정이다.
-
자주쓰이는 명령어 정리
-
1유형, 2유형 특징
-
1, 2, 3유형 모두 정리된 블로그
-
2유형 작성패턴 위주로 정리된 블로그
-
기출문제, 1, 2, 3유형 문제 모음
라이브러리
모델 학습, 적합
모델을 학습하고, 값을 예측할 때 공통적으로 작성하는 코드
model.fit(x_train, y_train)
y_train_predicted = model.predict(x_train)
y_test_predicted = model.predict(x_test)
라이브러리 호출 코드
-
LabelEncoder
from sklearn.preprocessing import LabelEncoder # label encoding # 범주형 데이터를 숫자로 변환 encoder = LabelEncoder() df["encoded"] = encoder.fit_transform(df["colName"]) # 여러 칼럼 인코딩 => 반복문 for col in cols : encoder = LabelEncoder() c_train[col] = encoder.fit_transform(c_train[col]) c_test[col] = encoder.fit_transform(c_test[col]) # one-hot encoding # 범주형 변수의 각 고유값을 이진 벡터로 변환([0, 0, 0, 1, 0, 0]) df = pd.get_dummies(df, columns = ["colName"]) # 전체 데이터 중 지정된 열(cols)만 원-핫 인코딩 # 다음과 같은 데이터가 있을 때, 'D'열을 제외하고 'A', 'B', 'C' 열만 인코딩함 c_train = pd.DataFrame({'A': ['apple', 'banana'], 'B': ['yes', 'no'], 'C': ['red', 'blue'], 'D' : ['trump', 'elon']}) cols = ['A', 'B', 'C'] c_train = pd.get_dummies(c_train, columns = cols) c_test = pd.get_dummies(c_test, columns = cols) # 결과 A_apple A_banana B_no B_yes C_blue C_red 0 1 0 0 1 0 1 1 0 1 1 0 1 0 -
train_test_split
from sklearn.model_selection import train_test_split x_train, x_test, y_train, y_test = train_test_split(x, y, test_size = 0.3) x_train, x_valid, y_train, y_valid = train_test_split(train.drop("charges", axis = 1), train["charges"], test_size = 0.14, random_state = 44) -
LinearRegression
from sklearn.linear_model import LinearRegression model = LinearRegression() model.fit(x_train, y_train) y_train_predicted = model.predict(x_train) y_test_predicted = model.predict(x_test) # y절편값 print(model.intercept_) # 각 독립변수의 회귀계수 print(model.coef_) -
RandomForestRegressor
from sklearn.ensemble import RandomForestRegressor # 트리 1000개 생성, 평가지표를 mae로 활용 model = RandomForestRegressor(n_estimators = 1000, criterion = "mae") -
GradientBoostingRegressor
from sklearn.ensemble import GradientBoostingRegressor model = GradientBoostingRegressor -
XGRegressor(Extreme Gradient Regressor)
from xgboost import XGRegressor -
평가지표(metrics)
# 연속형 종속변수 from sklearn.metrics import r2_score, mean_absolute_error, mean_squared_error # 범주형 종속변수 from sklearn.metrics import roc_auc_score, accuracy_score, precision_score, recall_score import numpy as np # 결정계수 print(r2_score(y_train, y_train_predicted)) # MAE print(mean_absolute_error(y_train, y_train_predicted)) # MSE print(mean_squared_error(y_train, y_train_predicted)) # RMSE print(np.sqrt(mean_squared_error(y_train, y_train_predicted))) # 범주형도 사용법 동일 -
의사결정나무 분류기, 랜덤포레스트 분류기
from sklearn.tree import DecisionTreeClassifier, RandomForestClassifier model = DecisionTreeClassifier() model = RandomForestClassifier() -
XGClassifier(Extreme Gradient Classifier)
from xgboost import XGBClassifier model = XGBClassifier(eval_metrics = "error", random_state = 100) -
서포트 벡터 분류기, 배깅 분류기, KNN 분류기, 다층 퍼셉트론 분류기
from sklearn.svm import SVC from sklearn.ensemble import BaggingClassifier from sklearn.neighbors import KNeighborsClassifier from sklearn.neural_network import MLPClassifier model = SVC() model = BaggingClassifier() model = KNeighborsClassifier() model = MLPClassifier() -
로지스틱 회귀
import sklearn.linear_model import LogisticRegression model = LogisticRegression() -
예측에 대한 확률값 계산
# 입력값을 넣었을 때 어떤 클래스에 속하는지 클래스를 직접 예측 y_test = model.predict(x_test) # 입력값을 넣었을 때 해당 입력값이 특정 클래스에 속할 확률 y_test_proba = model.predict_proba(x_test) -
표준화(스케일링)
from sklearn.preprocessing import StandardScaler, MinMaxScaler, RobustScaler scaler = standardScaler() # 평균 0, 표준편차 1인 표준정규분포 형식으로 변환 scaler = RobustScaler() # 중앙값, 사분위수를 활용해 상대적으로 이상치 영향 최소화 scaled_data = scaler.fit_transform(df) scaler = MinMaxScaler() # 최대 1, 최소 0에 맞춰 변환 cols = ["age", "bmi"] # fit_transform()은 2차원 DataFrame을 입력해야함 # 열이름 하나만 직접 넣을땐 train[["df"]] 이렇게 해야함 train[cols] = scaler.fit_transform(train[cols]) test[cols] = scaler.transform(test[cols])
1유형
# 파일 읽기
pd.read_csv()
# cp949는 csv 파일안에 데이터가 한글로 되어있을 경우
x_train = pd.read_csv("경로", encoding = "cp949")
################################
# 특정 열의 고유값 개수 반환
df["col"].nunique()
# 각 항목별 개수
df["col"].value_counts()
# 데이터를 csv파일로 저장
df.to_csv("output.csv", index = False)
# 비어있는 새 칼럼 추가
df["new"] = np.nan
# 데이터 추가
# 리스트 형태로 추가
df.loc["new"] = ["신메뉴", 500, 200, "특별"]
# 딕셔너리 형태로 행 추가
df.loc[5] = {"메뉴" : "아메리카노", "가격" : 4500, "칼로리" : 100}
################################
# 값 변경
df.replace("예전값", "새값")
# 결측치 채우기
# col열에 결측치를 모두 "값"으로 바꿈
df["col"] = df["col"].fillna("값")
## 범주형
# 최빈값 대체
mode_value = df["col"].mode()[0]
df["칼럼명"].fillna(mode_value, inplace = True)
# 특정 값으로 채두기
df["칼럼명"].fillna("특정값", inplace = True)
## 수치형
# 평균값 대체
df["칼럼명"].fillna(df["칼럼명"].mean(), inplace = True)
# 중앙값
df["칼럼명"].fillna(df["칼럼명"].median(), inplace = True)
################################
# null 개수
print(df.isnull().sum())
# null 비율
print(df.isnull().mean() * 100)
################################
# 값 삭제
# axis = 1 : 열(1이 길쭉하니까 열로 외우자)
df = df.drop("a", axis = 1, inplace = False) # a 칼럼 삭제, inplace = True : 원본 DataFrame이 변경됨
df = df.drop(1, axis = 0) # 인덱스가 1인 행 삭제
df = df.dropna() # 결측치가 하나라도 있는 행 삭제
df = df.dropna(subset = ["col"]) # col 열에 결측치가 있는 행만 삭제
# 여러 행, 열 삭제 => 리스트로 지정
# 인덱스 0, 2인 행 삭제
df = df.drop([0, 2], axis = 0)
# 'A', 'C' 열 삭제
df = df.drop(['A', 'C'], axis = 1)
# 열 'A'의 값이 2인 행 삭제
df = df[df['A'] != 2]
df = df.loc[df['A'] != 2]
################################
# 왜도, 첨도
v1 = df["col"].skew()
v2 = df["col"].kurt()
################################
df["col"] # 하나의 열에 해당하는 series가 출력
df[["col"]] # 열이 하나인 DataFrame
################################
# 인덱스 정렬
df = df.sort_index(ascending = False)
# 열 정렬(이름이 col인 열을 기준으로 정렬)
df = df.sort_values("col", ascending = True)
# 새로운 인덱스 생성
df = df.reset_index(drop = True)
# 특정 칼럼 기준 데이터프레임 정렬
df = df.sort_values("col", ascending = False)
# 여러 칼럼 기준으로 정렬할 때는 by를 꼭 써줘야함!
# 각각의 열에 대해 내림차순, 오름차순을 다르게 설정할 수도 있다.
df = df.sort_values(by = ["col1", "col2"], ascending = [True, False])
################################
# 평균으로부터 표준편차 * 1.5를 벗어나는 이상치의 합을 구하시오
std = df["age"].std() * 1.5
mean = df["age"].mean()
# 이상치의 조건
condition = (df["age"] > mean + std) | (df["age"] < mean - std)
# 이상치에 해당하는 age열을 구한뒤, 모두 더함
df.loc[condition]["age"].sum()
################################
# 문자열 어절 나누기
df["col"].str.split()
# 특정 문자 포함 여부
condition = df["col"].str.contains("특정문자")
df[condition]
################################
# object에서 날짜형 데이터로 변환
df["date"] = pd.to_datetime(df["date"], format = "%Y:%m:%d")
diff = df["100일"] - df["100시간"]
diff.dt.total_seconds()
diff.dt.total_seconds() / 60
diff.dt.total_seconds() / 60 / 60
2유형
### 데이터 탐색 및 초기처리
df.describe() # 수치형 칼럼의 통계값 반환
df.describe(include = "object") # 범주형 컬럼의 통계값 반환
df.describe(include = ["int64", "float64"]) # 특정 데이터 타입만 반환
################################
### 피쳐 엔지니어링
## 데이터 분리
# 수치형 데이터
numeric_data = df.select_dtypes(include = ["float64", "int64"])
# 범주형 데이터
categorical_data = df.select_dtypes(include = "object")
################################
# 중복값을 제거할 경우 기본적으로 먼저 나타난 값을 남겨둠.
# keep = "lost"를 설정 시 뒤의 값을 남겨둠
df = x_train.drop_duplicates(subset = ['A', 'B'], keep = "lost")
################################
### 모델링
## 분류
# 랜덤포레스트
from sklearn.ensemble import RandomForestClassifier
rf = RandomForestClassifier()
rf.fit(x_train[cols], y) # 지정된 열의 학습데이터와 정답 데이터를 학습
pred = rf.predict(x_test[col])
# 로지스틱 회귀
from sklearn.linear_model import LogisticRegression
model = LogisticRegression()
model.fit(x_train, y_train)
pred = model.predict(x_test)
################################
# lightgbm(Gradient Boosting Machine)
import lighgbm as lgb
model = lgb.LGBMClassifier() # 분류기
model.fit(x_train, y_train)
y_pred = model.predict(x_test)
model = lgb.LGBMRegressor() # 회귀모델
model.fit(x_train, y_train)
y_pred = model.predict(x_test)
2유형 공식
-
데이터 가져오기
-
결측치 확인 및 대체
-
라벨 인코딩(범주형 => 수치형)
-
원핫 인코딩(범주형 => 수치형)
3, 4에서 수치형 => 범주형일 경우 binning을 거쳐야 함(회귀문제)
-
파생변수 생성
-
스케일링(StandardScaler(범주형), MinMaxScaler(수치형))
-
데이터 분리(train_test_split)
-
모형학습(1. 단일모형 2. 앙상블 => RandomForest 많이 사용함)
-
모형 평가
-
하이퍼파라미터 튜닝
-
예측값 저장(index = False로 제출 형식에 맞춰야함)
-
read_csv로 저장된 데이터 확인
import pandas as pd
import numpy as np
### 1. 데이터 가져오기
# train, test가 분리 x
df = pd.read_csv("/data/train.csv")
# 분리 o
x_train = pd.read_csv("/data/x_train.csv", encoding = "cp949") # 한글인 경우
x_test = pd.read_csv("/data/x_test.csv")
y_train = pd.read_csv("/data/y_train.csv")
y_test = pd.read_csv("/data/y_test.csv")
print(df.shape) # shape는 메서드가 아님
print(df.info()) # 데이터 타입이 숫자형인지, 문자형인지 확인. 라벨(문자열을 0, 1, 2, 3... 그룹을 숫자로 표현), 원-핫 인코딩시 필수
print(df.describe()) # 기본적인 통계값
################################
### 2. 결측치 확인 및 대체
# 1. 전부 제거
# 2. 평균값, 중앙값으로 대체
# 3. 최빈값으로 대체
# 데이터가 쏠려있는 경우 중앙값을 많이 활용
print(df.isnull().sum()) # isna()나 isnull()이나 동일
# 중앙값으로 채우기
# 결측치를 포함하며 중복을 제외한 데이터의 종류를 ndarray로 반환
# 데이터
# name sex
# 0 Alice F
# 1 Bob M
# 2 Charlie M
# 3 David M
# 4 Eve F
# 5 Frank M
# 6 Grace F
# 7 Willy none
# unique()
# 결과 : ['F', 'M', nan]
df["sex"].unique()
# M 4
# F 3
# nan 1
df["sex"].value_counts() # 결측치 포함 하지 않으며 데이터 종류별 개수를 series로 반환
# 중앙값으로 대체
missing = ["bill_length_mm", "bill_depth_mm", "flipper_length_mm", "body_mass_g"]
for i in missing :
df[i] = df[i].fillna(df[i].median())
df["sex"] = df["sex"].fillna("male")
# 결측치 채우진거 확인
# 둘중에 하나 실행해서 확인
df.isna().sum()
df.info()
################################
### 3. 라벨 인코딩
from sklearn.preprocessing import LabelEncoder
encoder = LabelEncoder()
label = ["species", "island", "sex"]
df[label] = df[label].apply(encoder.fit_transform)
# 열별로 하나씩 라벨인코딩 적용
from sklearn.preprocessing import LabelEncoder
encoder = LabelEncoder()
print(encoder.fit_transform(x["am"])) # am열만 라벨인코딩
################################
### 4. 원핫 인코딩
import pandas as pd
category = ["island", "sex"]
for i in category :
df[i] = df[i].astype("category")
df = pd.get_dummies(df)
################################
### 5. 파생변수 생성
# qcut() : 분위수 기반으로 구간화 & 파생변수 생성
df["body_mass_g_qcut"] = pd.qcut(df["body_mass_g"], 5, labels = False)
df["body_mass_g_qcut"].value_counts() # 5구간으로 나눈 위, 확인
################################
### 6. 스케일링
from sklearn.preprocessing import MinMaxScaler
scale_list = ["bill_length_mm", "bill_depth_mm", "flipper_length_mm", "body_mass_g"]
scaler = MinMaxScaler()
df[scale_list] = scaler.fit_transform(df[scale_list])
df.head()
################################
### 7. 데이터 분리
x = df.iloc[:,1:] # species 열을 제외한 나머지(학습데이터)
y = df["species"] # species 열(정답 데이터)
print
from sklearn.model_selection import train_test_split
# stratify 속성 : species 열을 기준으로 층화추출. 안해줘도 나오긴 하지만 설정을 하면 성능이 더 높아짐
x_train, x_test, y_train, y_test = train_test_split(x, y, test_size = 0.2, random_state = 1, stratify = df["species"])
################################
### 8. 모형학습
# 첫번째 모형
from sklearn.ensemble import RandomForestClassifier
model1 = RandomForestClassifier()
model1.fit(x_train, y_train)
pred = model1.predict(x_test)
print(pred)
# 두번째 모형
from sklearn.ensemble import AdaBoostClassifier
model2 = AdaBoostClassifier()
model2.fit(x_train, y_train)
pred = model2.predict(x_test)
print(pred)
# 세번째 모형
from lightgbm import LGBMClassifier
model3 = LGBMClassifier()
model3.fit(x_train, y_train)
pred = model3.predict(x_test)
print(pred)
# 앙상블
from sklearn.ensemble import VotingClassifier
# soft : 확률, hard : 정수형으로 투표 결과 반영
clf = VotingClassifier(estimators = [("rf", model1), ("ad", model2)], voting = "hard")
clf.fit(x_train, y_train)
pred4 = clf.predict(x_test)
################################
### 9. 모형평가
from sklearn.metrics import accuracy_score, roc_auc_score, classification_report
print("랜덤포레스트 정확도", accuracy_score(y_test, pred1))
print("AdaBoost 정확도", accuracy_score(y_test, pred2))
print("LightGBM 정확도", accuracy_score(y_test, pred3))
print("HardVoting 정확도", accuracy_score(y_test, pred4))
################################
### 10. 하이퍼파라미터 튜닝
from sklearn.model_selection import GridSearchCV
params = {"n_estimators" : [50, 100], "max_depth" : [4, 6]}
model5 = RandomForestClassifier()
clf = GridSearchCV(estimator = model5, param_grid = params, cv = 3)
clf.fit(x_train, y_train)
print(clf.best_params_)
################################
### 11. 예측값 저장(index = False로 제출해야 한다)
pd.DataFrame({"id" : y_test.index, "pred" : pred3}).to_csv("/content/00300.csv", index = False)
################################
### 12. read_csv로 저장된 데이터 폼 확인
final = pd.read_csv("/content/00300.csv")
print(final)
3유형
t_test
| 단일표본검정 | 모집단 1개 | 한그룹 | 과자무게가 200g에서 달라졌는지 |
|---|---|---|---|
| 대응(쌍체)표본검정 | 모집단 2개(같은 집단) | 같은 그룹 | 신약 효과 검정(사전/사후) |
| 독립표본검정 | 모집단 2개 | 서로 다른 그룹 | 1반과 2반의 성적 차이 |
-
단일표본검정
shapiro-wilk를 수행 후, 정규분포를 따르면 t-test, 따르지 않으면 wilcoxon(비모수검정) 검정을 수행
from scipy import stats stats.ttest_1samp(칼럼명, 검증값, alternative)1) 모집단에 대해 정규분포 검정 : shapiro-wilk
귀무가설 : 데이터가 정규분포 따른다
대립가설 : 따르지 않는다
p-value : p >= 0.05 : 귀무가설 채택(정규분포 따른다)
from scipy import stats stat, p_value = stats.shapiro(df["무게"]) # p-value가 0.05보다 작으면 기각 => 정규분포 따르지 않는다 # wilcoxon 부호순위검정 사용 print(f"shapiro-wilk test statistic : {stat}, p-value : {p_value}")2) 정규성 검정되지 않았을 경우 : wilcoxon
목적 : 정규성을 따르지 않는 데이터의 중앙값을 특정 값과 비교
가정 : 비교하려는 두 값은 대칭적으로 분포한다고 가정
귀무가설 : 데이터의 중앙값은 기준값과 같다
대립가설 : 데이터의 중앙값은 기준값과 같지 않다
from scipy import stats # alternative # less 귀무가설 : df["무게"] - 120 >= 0 # greater 귀무가설 : df["무게"] - 120 <= 0 # two-sided 귀무가설 : df["무게"] - 120 == 0 stat, p_value = stats.wilcoxon(df["무게"] - 120, alternative = "less") print(f"wilcoxon test statistic : {stat}, p-value : {p_value}") # p_value 해석 # p <= 0.05 => 귀무가설 기각 => df["무게"]가 120보다 작다3) 정규성을 따르는 경우 : 단일 표본 t-검정
목적 : 정규성을 따르는 데이터의 평균을 특정 값과 비교
-
two-sided
H0 : 모집단 평균은 특정 값과 같다
-
greater
H0 : 모집단 평균은 특정 값보다 작거나 같다
-
less
H0 : 모집단 평균은 특정 값보다 크거나 같다
from scipy import stats stat, p_value = stats.ttest_1samp(df["무게"], 120) -
-
대응 표본 검정
같은 집단의 전, 후 비교
1) 정규성 검정 : shapiro-wilk
차이값의 정규성 검토
H0 : 차이값이 정규분포를 따른다
from scipy import stats df["diff"] = df["before"] - df["after"] stat, p_value = stats.shapiro(df["diff"]) # p_value 해석 # p_value <= 0.05 : 귀무가설 기각 => 차이값은 정규분포를 따르지 않는다2) 정규성을 따르지 않는 경우 : wilcoxon
from scipy import stats stat, p_value = stats.wilcoxon(df["before"], df["after"], alternative = "greater") # p_value 해석 # p_value <= 0.05 : 귀무가설 기각 # greater => 첫번째 데이터가 두번째 데이터보다 크다 # less => 첫번째 데이터가 두번째 데이터보다 작다3) 정규성을 따르는 경우 : 단일표본검정
-
two-sided
H0 : 사전-사후 평균의 차이가 0이다
-
greater
H0 : 사전-사후 평균의 차이가 0 이하이다
-
less
H0 : 사전-사후 평균의 차이가 0 이상이다
from scipy import stats stat, p_value = stats.ttest_rel(df["before"], df["after"], alternative = "less") # p <= 0.05 : 귀무가설 기각 => before와 after간의 차이가 있다. -
-
독립표본검정
목적 : 서로 다른 두 집단의 평균이 동일한지 확인
예 : 신약 투여 집단과 대조 집단의 혈압 비교, 두 클래스 학생들의 시험 점수비교
조건 : 두 집단이 서로 독립, 정규성 및 등분산성 여부에 따라 적합한 검정 선택
1) 모집단에 대해 정규성 검정 : shapiro-wilk
- H0 : 각 집단의 데이터가 정규분포를 따른다
from scipy import stats # 귀무가설 : 정규성을 따른다 stat_A, p_value = stats.shapiro(A) stat_B, p_value = stats.shapiro(B) # p_value <= 0.05이면 귀무가설 기각 => 정규성을 따르지 않는다2-1) 정규성 검정 x => mann-whitney u 검정
-
two-sided
H0 : 두 집단의 중앙값의 차이가 0이다
-
greater :
H0 : 두 집단의 중앙값의 차이가 0 이하이다
-
less :
H0 : 두 집단의 중앙값의 차이가 0 이상이다
# 귀무가설 : 두 집단의 중앙값이 동일하다 stat, p_value = stats.mannwhitneyu(A, B, alternative = "less") # p_value < 0.05이면 귀무가설 기각 => 두 집단의 중앙값이 다르다2-2) 정규성 따름 => 등분산 검정
- H0 : 두 집단의 분산이 동일하다
# 귀무가설 : 두 집단의 분산이 동일하다 stat, p_value = stats.levene(A, B) # p_value < 0.05이면 귀무가설 기각 => 두 집단의 분산이 다르다3-1) 등분산성 o => t-test
-
two-sided :
H0 : 두 집단의 평균의 차이가 0이다
-
greater :
H0 : 두 집단의 평균의 차이가 0 이하이다
-
less :
H0 : 두 집단의 평균의 차이가 0 이상이다
# 귀무가설 : 두 집단의 평균은 동일하다 # 대립가설 : # "two-sided" : 두 집단 평균은 다르다 # "greater" : A가 B보다 크다 # "less" : A가 B보다 작다3-2) 등분산성 x => t-test with equal_var = False
stat, p_value = stats.ttest_ind(A, B, equal_var = False, alternative = "less")
범주형 데이터 분석(카이제곱)
-
적합도 검정
관찰값, 기대값 사이의 차이를 검정하여 데이터가 특정 분포에 적합한지를 확인한다
H0 : 관찰값과 기대값의 차이가 0이다(주어진 데이터가 특정 분포에 적합하다)
from scipy.stats import chisquare observed = [50, 30, 20] expected = [40, 40, 20] stat, p_value = chisquare(f_obs = observed, f_exp = expected) if (p_value < .05) : print("귀무가설 기각 : 관찰값과 기대값 사이에 차이가 있다") else : print("귀무가설 채택 : 관찰값과 기대값 사이에 차이가 없다") -
독립성 검정
두 범주형 변수 간의 독립성 확인
H0 : 두 변수는 서로 독립적이다
from scipy.stats import chi2_contingency import pandas as pd data = { "성별" : ["남", "여", "남", "여", "남", "여"] "반응" : ["긍정", "부정", "긍정", "긍정", "부정", "부정"] } table = pd.crosstab(df["성별"], df["반응"]) print("교차표 : \n", table) stat, p_value, dof, expected = chi2_contingency(table) if p_value <= .05 : print("귀무가설 기각 : 두 변수는 독립적이지 않다") else : print("귀무가설 채택 : 두 변수는 독립적이다") -
동질성 검정
여러개의 집단이 동일한 분포를 따르는지 확인.
H0 : 모든 집단은 동일한 분포를 따른다
data = { "집단" : ["A", "A", "A", "B", "B", "B", "C", "C", "C"], "반응" : ["긍정", "부정", "긍정", "긍정", "긍정", "부정", "부정", "부정", "긍정"] } df = pd.DataFrame(data) table = pd.crosstab(df["집단"], df["반응"]) print(table) stat, p_value, dof, expected = chi2_contingency(table) if p_value <= .05 : print("귀무가설 기각 : 집단들은 동일한 분포를 따르지 않는다") else : print("귀무가설 채택 : 집단들은 동일한 분포를 따른다")
회귀분석
-
상관계수
df.corr() # 피어슨 상관계수(default) df.corr(method = "spearman") # 스피어만 상관계수 df.corr(method = "kendall") # 켄달카우 -
상관계수 검정
H0 : 두 변수간에 상관관계가 없다
from scipy import stats stats.pearsonr(x, y) stats.spearmanr(x, y) stats.kendalltau(x, y) -
단순 선형 회귀분석
OLS(Ordinary Least Squares) : 최소제곱법
from statsmodels.formula.api import ols model = ols("키 ~ 몸무게", data = df).fit() # 결정계수 model.rsquared # 기울기 model.params["몸무게"] # 절편 model.params["Intercept"] # p-value model.pvalues["몸무게"] # 예측값 newdata = pd.DataFrame({"몸무게" : [50]}) model.predict(newdata) # 예측값과 예측값에 대한 신뢰구간, 예측구간 newdata = pd.DataFrame({"몸무게" : [50]}) pred = model.get_prediction(newdata) pred.summary_frame(alpha = 0.05) # 95% ### 실행결과 이미지 참고 ### # 잔차제곱합 df["잔차"] = df["키"] - model.predict(df["몸무게"]) (df["잔차"] ** 2).mean()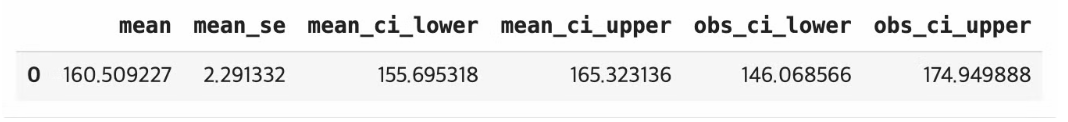
-
다중선형회귀분석
# 잔차 구하는 메서드 model.resid # 광고비, 플랫폼 회귀계수의 95% 구간 # 광고비의 회귀계수가 [0.1, 0.3] => 광고비가 1 증가할 때 종속변수의 변화량이 95% 신뢰수준에서 0.1~0.3 사이에 있다 model.conf_int(alpha = .05) # confidenct interval newdata = pd.DataFrame({ "광고비" : [50], "플랫폼" : [20] }) pred = model.get_prediction(newdata) pred.summary_frame(alpha = .05)
분산분석(ANOVA)
-
일원 분산분석(one-way ANOVA)
1) shapiro-wilk
print(stats.shapiro(df['A'])) print(stats.shapiro(df['B'])) print(stats.shapiro(df['C'])) print(stats.shapiro(df['D']))2-1) 1에서 정규분포 따르지 않으면 kruskal-wallis
stats.kruskal(df['A'], df['B'], df['C'], df['D'])2-2) 정규성 따르면 등분산 검정
print(stats.levene(df['A'], df['B'], df['C'], df['D']))3) 일원분산분석 : f_oneway
print(stats.f_oneway(df['A'], df['B'], df['C'], df['D']))4) 사후검정 : tukey, bonferroni
from scipy import stats from statsmodels.stats.multicomp import pairwise_tukeyhsd, MultiComparison tukey_result = pairwise_tukeyhsd(df_melt["value"], df_melt["variable"], alpha = .05) print(tukey_result.summary()) mc = MultiComparison(df_melt["value"], df_melt["variable"]) bon_result = mc.allpairtest(stats.ttest_ind, method = "bonf") print(bon_result[0])이원 분산 분석(two-way ANOVA)
두개의 요인에 따라 평균의 차이 검정
from statsmodels.formula.api import ols from statsmodels.stats.anova import anova_lm # 토마토수 : 종속변수 # C() : 범주형 변수 지정 # 종자 : 토마토 수에 영향을 미칠것으로 예상되는 종자 종류 # C()로 감싸지 않으면 변수는 숫자로 처리됨 # C(종자):C(비료) : 특정 종자와 특정 비료 조합이 토마토 수에 미치는 영향 분석 # 두 변수간의 상호작용 효과를 모델에 포함한다. model = ols('토마토수 ~ C(종자) + C(비료) + C(종자):C(비료)', data = df).fit() anova_lm(model)
최적화
시간 여유가 있으면 하이퍼파라미터 튜닝을 진행
-
랜덤포레스트
max_depth : 3 ~ 12
n_estimators : 100, 200, 400, 800, 1000
-
XGBoost
max_depth : 3 ~ 12
n_estimators : 100 ~ 1000
learning_rate : 0.01 ~ 0.1(n_estimators 값과 반비례)
댓글남기기